La traduction des gènes, et particulièrement la phase d’initiation, est une étape importante de la régulation de l’expression génique qui est longtemps restée méconnue. Elle permet entre autre à la cellule de réagir très rapidement à un stimulus en activant la traduction d’ARN messagers accumulés sous forme inactive. Citons, par exemple, l’activation des ARNm maternels lors de la fécondation des ovocytes, la réponse à l’interféron gamma lors d’une infection virale, ou l’activation de la traduction de certains ARNm lors du processus de mémorisation. Cette étape précoce qu’est l’initiation est la cible de nombreuses régulations. Notre équipe s’intéresse aux mécanismes moléculaires gouvernant l’initiation de la traduction, et plus particulièrement à l’influence de la structure des ARNm sur ce processus. Notre modèle d’étude est la traduction des virus, et plus particulièrement celle du virus HIV. Nous cherchons à définir les spécificités de la traduction virale afin de définir de nouvelles cibles thérapeutiques.
Le mécanisme d’initiation de la traduction en quelques mots :
L’initiation de la traduction chez les eucaryotes débute par l’attachement d’un complexe comprenant la petite sous-unité ribosomale (40S) sur la coiffe, à l’extrémité 5’ de l’ARNm et d’au minimum une dizaine de facteurs protéiques connus sous le nom de « facteurs d’initiation » (ou eIF pour « eukaryotic Initiation Factors »). Ce complexe progresse ensuite le long de la région 5’ non traduite (5’UTR) jusqu’à atteindre le codon d’initiation (étape de « balayage ») où le ribosome complet (80S) sera assemblé. Au cours de ce processus, chacun des facteurs d’initiation à un rôle précis qui permet au final de positionner un ribosome compétent pour la traduction sur le « bon » codon d’initiation. Ce mécanisme est connu sous le nom d’initiation dépendante de la coiffe.


initiation canonique de la traduction dite « Coiffe-dépendante
L’entrée interne des ribosomes :
Cependant, dans un certain nombre d’exception, la 5’UTR a la propriété d’attirer directement le complexe ribosomal indépendamment de la présence d’une coiffe. La 5’ UTR est alors qualifiée de Site d’Entrée Interne des Ribosomes gènes spécifiques peut continuer, même si certains facteurs d’initiation sont altérés de manière physiologique (régulation générale de la traduction) ou pathologique (suite à un stress). C’est ainsi que des génomes viraux sont toujours traduits bien que les virus correspondants aient préalablement endommagé le système de traduction cellulaire.


Les différents types d’IRES et les facteurs d’initiation requis.
La traduction du VIH :
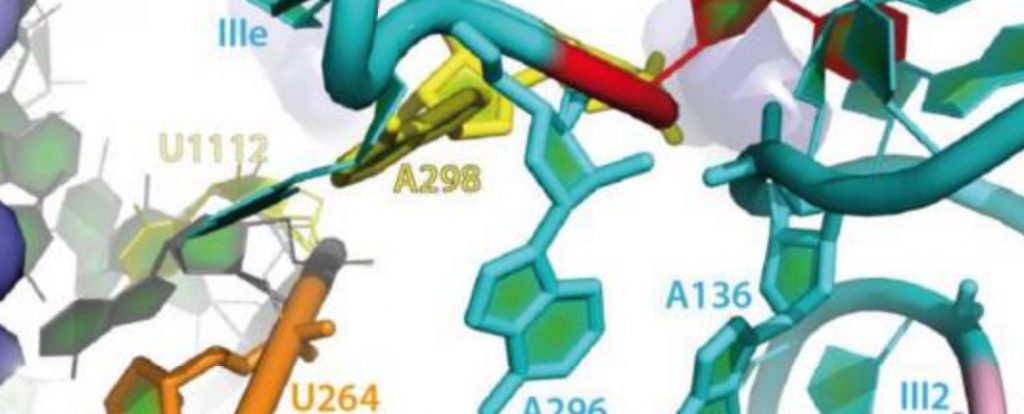
Notre équipe s’intéresse plus particulièrement à la traduction de la polyprotéine Gag du virus VIH responsable du S.I.D.A. Ce virus est traduit grâce à une combinaison des mécanismes coiffe-dépendant et d’entrée interne des ribosomes (Balvay et al, 2007). Nous avons montré que ce virus à la caractéristique unique de posséder une IRES à l’intérieur de la séquence codant pour le gène Gag (Herbreteau et al, 2005). Cette propriété est dépendante de la structure secondaire adoptée par les 500 premiers nucléotides de l’ARN de la région codante. Cette structure est conservée chez les lentivirus de primates (Weill et al, 2010). Elle permet de recruter directement la petite sous unité du ribosome et le facteur central d’initiation eIF3 (Chamond et al, 2010 ; Locker et al, 2011).


Modèle de la structure secondaire des régions codantes du gène gag des lentivirus de primates
Il semble donc que l’ARN génomique de ces virus puisse être traduit de manière classique, mais aussi par entrée interne des ribosomes soit dans la 5’UTR soit directement dans la région codante (Balvay et al, 2007 ; Chamond et al, 2010 ; de Breyne et al, 2012).


Différents modes d’initiation de la traduction de Gag
L’entrée interne des ribosomes dans la 5’UTR est particulièrement active durant la phase G2/M du cycle cellulaire, ceci grâce à la fixation de protéines cellulaires sur la 5’UTR (Vallejos et al, 2012 ; Vallejos et al, 2011). L’ensemble de ces propriétés pourrait permettre de réguler la production de Gag au cours du cycle viral.


Les spécificités structurales de la traduction virale comme cible thérapeutique :
Nous étudions les mécanismes moléculaires de ce mode d’initiation et cherchons à définir le rôle de la structure d’ARN. Pour sonder la structure des ARN dans de nombreuses conditions, étudier l’impact des mutations sur cette structure, ou définir les sites d’interaction ARN-protéine, nous nous sommes dotés d’un système d’étude de la structure secondaire des ARN semi-automatisé utilisant un séquenceur capillaire ( SHAPE , DMS, CMCT, sondes enzymatiques). Ceci nous a permis entre autre d’identifier de nouveaux déterminants de la dimérisation du génome du VIH (Deforges et al, 2012) ou de modéliser la structure de la 5’UTR de l’homologue du VIH infectant les félins (James & Sargueil, 2008). Pour étudier en détail les mécanismes moléculaires, nous reconstituons entièrement le processus d’initiation in vitro , à partir des constituants purifiés (10 facteurs d’initiation, les sous-unités ribosomales, le tRNAMet, protéines accessoires). Ceci nous permet d’étudier la dynamique structurale des évènements menant à la formation du complexe d’initiation. Les virus sont un exemple formidable de la compaction de l’information, ainsi leurs ARN possède à la fois des propriétés codantes et des propriétés structurale lui conférant d’autres fonctions. Nous cherchons à définir la spécificité des structures impliqués dans la traduction afin de les utiliser comme cibles thérapeutiques. Pour valider cette approche nous criblons une banque de petites molécules variées afin d’en isoler une capable d’inhiber spécifiquement la traduction d’un virus donné.
– Publications :
Balvay L, Lastra ML, Sargueil B, Darlix JL, Ohlmann T (2007) Translational control of retroviruses. Nat Rev Microbiol 5: 128-140
Chamond N, Locker N, Sargueil B (2010) The different pathways of HIV genomic RNA translation. Biochem Soc Trans 38: 1548-1552
de Breyne S, Chamond N, Decimo D, Trabaud MA, Andre P, Sargueil B, Ohlmann T (2012) In vitro studies reveal that different modes of initiation on HIV-1 mRNA have different levels of requirement for eIF4F. The FEBS journal 279: 3098-3111
Deforges J, Chamond N, Sargueil B (2012) Structural investigation of HIV-1 genomic RNA dimerization process reveals a role for the Major Splice-site stem loop. Biochimie 94: 1481-1489
Herbreteau CH, Weill L, Decimo D, Prevot D, Darlix JL, Sargueil B, Ohlmann T (2005) HIV-2 genomic RNA contains a novel type of IRES located downstream of its initiation codon. Nat Struct Mol Biol 12: 1001-1007
James L, Sargueil B (2008) RNA secondary structure of the feline immunodeficiency virus 5’UTR and Gag coding region. Nucleic Acids Res 36: 4653-4666
Locker N, Chamond N, Sargueil B (2011) A conserved structure within the HIV gag open reading frame that controls translation initiation directly recruits the 40S subunit and eIF3. Nucleic Acids Res 39: 2367-2377
Vallejos M, Carvajal F, Pino K, Navarrete C, Ferres M, Huidobro-Toro JP, Sargueil B, Lopez-Lastra M (2012) Functional and structural analysis of the internal ribosome entry site present in the mRNA of natural variants of the HIV-1. PLoS One 7: e35031
Vallejos M, Deforges J, Plank TD, Letelier A, Ramdohr P, Abraham CG, Valiente-Echeverria F, Kieft JS, Sargueil B, Lopez-Lastra M (2011) Activity of the human immunodeficiency virus type 1 cell cycle-dependent internal ribosomal entry site is modulated by IRES trans-acting factors. Nucleic Acids Res 39: 6186-6200
Weill L, James L, Ulryck N, Chamond N, Herbreteau CH, Ohlmann T, Sargueil B (2010) A new type of IRES within gag coding region recruits three initiation complexes on HIV-2 genomic RNA. Nucleic Acids Res 38: 1367-1381